以H7N9的數據作為例子吧!
setwd("設定路徑-記得是: /")
#使用epitools內的epicurve.weeks或epicurve.months等, 其實還有很多的, 可在RStudio內點epitools選...
library(epitools)
#使用讀入Excel的功能
library(readxl)
h7n9<-read_excel("文件名.xls")
h7n9$onset_date<-as.Date(h7n9$onset_date)
h7n9.week<-epicurve.weeks(h7n9$onset_date,format="%y-%m-%d",axisnames=F,legend="H7N9
week cases",xlab="Weeks",ylab="Cases
no",col="green",segments=TRUE)
#format="%y-%m-%d" 時間變項的格式,
#axisnames=F要X軸的名義, 但我不要, 因為顯示出來的是一串數字, 不是週數,
#legend="H7N9 week cases",
#xlab="Weeks",
#ylab="Cases no",
#col="green",
#segments=TRUE 是否要以每細格代表1個案
#設定X軸, 這樣會好睇D!
axis(1,at=h7n9.week$xvals,labels=h7n9.week$cweek)
#設定圖題
title("H7N9週流行曲線")
參考資料: http://www.elsenburg.com/vetepi/BPEL/BPEL_2014_05_EpidemicCurve.pdf
[更新: 2016/1/1]
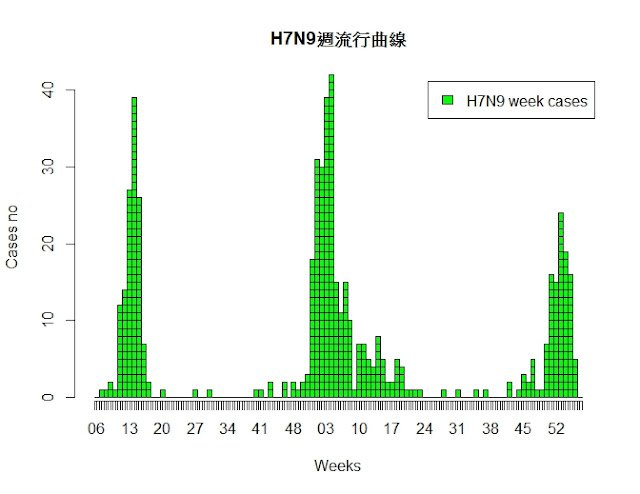 |
| 完成作品,都不錯的... |
h7n9<-read.csv("H7N920160115.csv",header = T)
h7n9$onset_date<-as.Date(h7n9$onset_date)
library(epitools)
h7n9.week<-epicurve.weeks(h7n9$onset_date, format = "%Y/%m/%d",segments=TRUE,strata = h7n9$infect_pro,xlab="Weeks",col=h7n9$infect_pro,axisnames=F,main="H7N9 update to 2016/1/1")
axis(1,at=h7n9.week$xvals,labels=paste(h7n9.week$cyear,h7n9.week$cweek,sep = "-"))
h7n9$onset_date<-as.Date(h7n9$onset_date)
library(epitools)
h7n9.week<-epicurve.weeks(h7n9$onset_date, format = "%Y/%m/%d",segments=TRUE,strata = h7n9$infect_pro,xlab="Weeks",col=h7n9$infect_pro,axisnames=F,main="H7N9 update to 2016/1/1")
axis(1,at=h7n9.week$xvals,labels=paste(h7n9.week$cyear,h7n9.week$cweek,sep = "-"))

沒有留言:
張貼留言